A
M 1 2 3 4 5 6 7 8 9 10 111213141516CM
.
-
-
w_,
•
•
-
-
--~
--
--~-
-
..
_
B
(::~)/'
-21M13primer
c
Exon
14
lntron 14
A.A.G ~ TT A.GGNNNN T C .).G C T MNN N GNGNG
Wildtype
r
taaatcagctgagtgtgtg
5' AAGCTIAGg
L
atttgctgagtgtgtgaac
Mutant
Fig.
2.
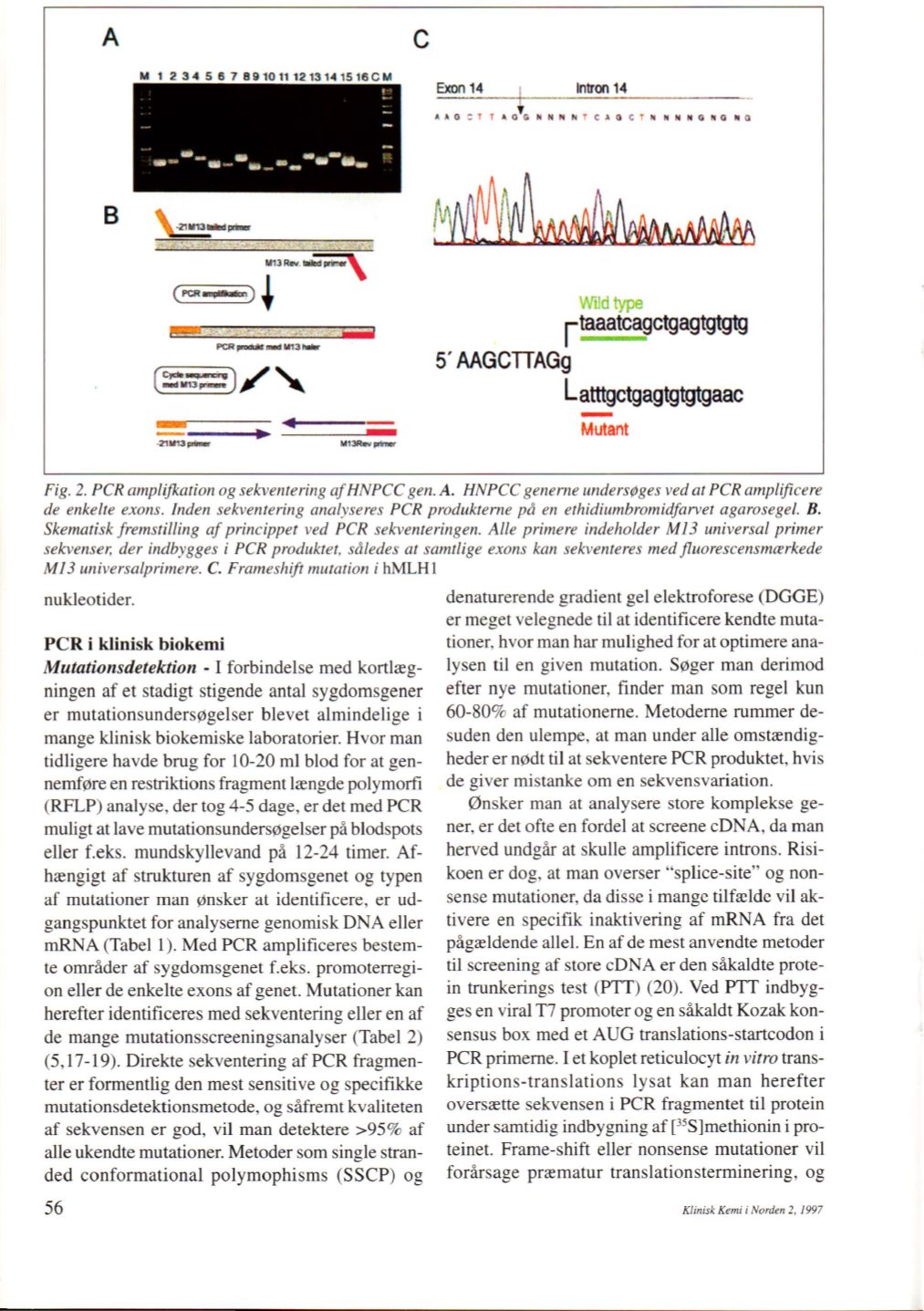
PCRamplifkation og sekventeringafHNPCCgen. A. HNPCCgenerne underspges vedat PCRamplificere
de enkelte exons. lnden sekventering analyseres PCR produkterne på en ethidiumbromidfarvet agarosegeL B.
Skematisk fremstilling af princippet ved PCR sekventeringen. Alle primere indeholder M13 universal primer
sekvenser, der indbygges
i
PCR produkter, således at samttige exons kan sekventeres med fluorescensmcerkede
MJ3 universalprimere.
C.
Frameshift mutation
i
hMLHl
nukleotider.
PCR i klinisk biokemi
Mutationsdetektion
-I
forbindelse med kortl:eg–
ningen af et stadigt stigende antal sygdomsgener
er mutationsunderss>)gelser blevet almindelige i
mange klinisk biokemiske laboratorier.
H
vor man
tidligere havde brug for 10-20mi blod for at gen–
nemfs>)re en restriktions fragment l:engde polymorft
(RFLP) analyse, der tog4-5 dage, erdet med PCR
muligt at lavemutationsunderss>)gelserpåblodspots
eller f.eks. mundskyllevand på 12-24 timer.
Af–
h:engigt af strukturen af sygdomsgenet og typen
af mutationer man S')nsker at identificere, er ud–
gangspunktet for analyserne genomiskDNA eller
mRNA (Tabel 1). Med PCR amplificeres bestern–
te ornråder af sygdomsgenet f.eks. promoterregi–
on eller de enkelte exons afgenet. Mutationer kan
herefter identificeresmed sekventering eller en af
de mange mutationsscreeningsanalyser (Tabel 2)
(5,17-19). Direkte sekventering afPCR fragmen–
ter er formenttig den mest sensitive og specifikke
mutationsdetektionsmetode, og såfremt kvaliteten
af sekvensen er god, vil man detektere >95% af
alle ukendtemutationer.Metoder som single stran–
ded conformational polymophisms (SSCP) og
56
denaturerende gradient gel elektroforese (DGGE)
ermeget velegnede til at identificerekendtemuta–
tioner, hvorman harmulighed for at optimere ana–
lysen til en given mutation. SS')ger man derimod
efter nye mutationer, finder man som regel kun
60-80% af mutationeme. Metodeme rummer de–
suden den ulempe, at man under alle omstrendig–
hederer ns>)dt til at sekventerePCRproduktet, hvis
de giver mistanke om en sekvensvariation.
0nsker man at analysere store komplekse ge–
ner, er det ofte en fordel at screene cDNA, daman
herved undgår at skulle amplificere introns. Risi–
koen er dog, at man overser "splice-site" og non–
sensemutationer, da disse imange tilf:elde vil ak–
tivere en specifik inaktivering af mRNA fra det
påg:eldende alle!. En af demest anvendtemetoder
til screening af store cDNA er den såkaldte prote–
in trunkerings test (PTT) (20). Ved PTT indbyg–
ges envira]T7 promoterogen såkaldtKozak kon–
sensus box med et AUG translations-startcodon i
PCRprimerne.
I
et koplet reticulocyt
in vitro
trans–
kriptions-transtations lysat kan man herefter
overs:ette sekvensen i PCR fragmentet til protein
under samtidig indbygning af
[3
5
S]methionin ipro–
teinet. Frame-shift eller nonsense mutationer vil
forårsage pr:ematur translationsterminering, og
Klinisk Kemi
i
Norden 2, 1997


